💡
原文中文,约2400字,阅读约需6分钟。
📝
内容提要
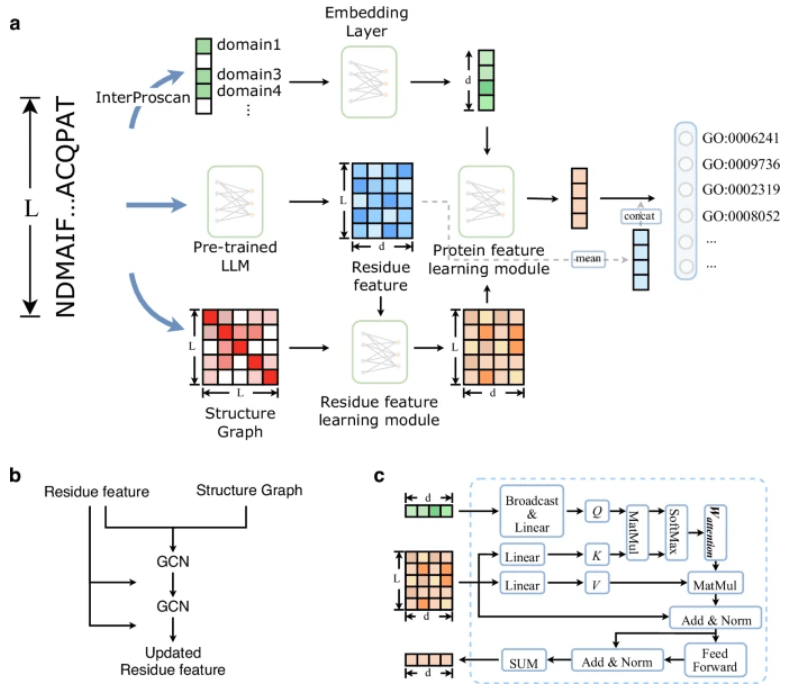
中南大学团队提出了一种名为DPFunc的深度学习方法,利用结构域信息进行蛋白质功能预测。DPFunc通过残基特征学习和图神经网络,准确识别关键区域,尤其在稀有功能和低序列相似性蛋白质中表现优越。该研究成果发表于《Nature Communications》。
🎯
关键要点
- 中南大学团队提出了一种名为DPFunc的深度学习方法,用于蛋白质功能预测。
- DPFunc利用结构域信息,能够准确识别蛋白质结构中的关键区域,尤其在稀有功能和低序列相似性蛋白质中表现优越。
- 该研究成果发表于《Nature Communications》,标题为《DPFunc: accurately predicting protein function via deep learning with domain-guided structure information》。
- DPFunc由三个模块组成:残基级特征学习模块、蛋白质水平特征学习模块和蛋白质功能预测模块。
- 模型通过预训练的蛋白质语言模型生成初始特征,并使用图神经网络更新和学习残基层特征。
- DPFunc在预测具有高IC值特征的GO项时表现出色,尤其在样本较少的情况下优于其他方法。
- 通过替换域注意力块,团队证明了结构域信息在DPFunc中的关键作用,显著提高了模型性能。
- DPFunc能够在没有序列相似性的情况下,准确捕捉结构相似性并预测功能,显示出其在蛋白质功能预测方面的潜力。
- DPFunc有效检测酶功能的重要活性位点,但在无序区域寻找活性位点仍然是一个挑战。
- 未来的挑战包括如何准确预测蛋白质在动态环境中的功能变化。
➡️


